La artrosis (OA) es una enfermedad compleja en la que diferentes factores ambientales interactúan con múltiples factores genéticos. Esta revisión se centra en los estudios que han contribuido a descubrir los factores genéticos de susceptibilidad a la OA. También se tratan con detalle los loci más relevantes en la actualidad, como GDF-5, el locus en el cromosoma 7q22, MCF2L, DOT1L, NCOA3 y los provenientes del estudio arcOGEN. Además, se discuten las diferentes aproximaciones que pueden servir para minimizar los problemas específicos del estudio de la genética de la OA. Entre ellas se encuentran la estandarización de los fenotipos, el estudio de microsatélites y también el uso de otras estrategias de estudio, como metaanálisis de GWAS y análisis basados en genes. Mediante estos nuevos enfoques se espera contribuir al descubrimiento de nuevos factores genéticos de susceptibilidad a la OA.
Osteoarthritis (OA) is a complex disease caused by the interaction of multiple genetic and environmental factors. This review focuses on the studies that have contributed to the discovery of genetic susceptibility factors in OA. The most relevant associations discovered until now are discussed in detail: GDF-5, 7q22 locus, MCF2L, DOT1L, NCOA3 and also some important findings from the arcOGEN study. Moreover, the different approaches that can be used to minimize the specific problems of the study of OA genetics are discussed. These include the study of microsatellites, phenotype standardization and other methods such as meta-analysis of GWAS and gene-based analysis. It is expected that these new approaches contribute to finding new susceptibility genetic factors for OA.
Los primeros estudios que han permitido mostrar el componente genético de la artrosis (OA, osteoarthritis) han sido los estudios de gemelos, de riesgo relativo entre hermanos y de agregación familiar. Todos estos trabajos permiten obtener una estimación de la heredabilidad, que cuantifica la importancia de los factores genéticos en la enfermedad. Las estimaciones de heredabilidad en la OA varían en función de los diferentes estudios consultados. Sin embargo, se puede decir que la heredabilidad de la OA de rodilla es del 40%, la de la mano se sitúa en torno al 65% y la de OA de cadera alrededor del 60%. La OA de columna presenta la heredabilidad más elevada, del 70%1. Estos trabajos han dado paso a otros estudios orientados a descubrir los factores genéticos de susceptibilidad a OA.
Estudios de ligamientoLos estudios de ligamiento evalúan la cosegregación de marcadores genéticos con la enfermedad en familias con múltiples casos de OA. El ligamiento se observa cuando varios familiares afectos comparten la misma variante alélica para un marcador. Una vez localizada la región genética que segrega con la enfermedad, esta se satura con más marcadores hasta que la región de ligamiento se acorte y los genes se puedan priorizar por su función. Los estudios de ligamiento en OA se llevaron a cabo en diferentes cohortes con distinto fenotipo. En OA de mano se realizaron los estudios de Framingham (EE.UU.), Islandia y Finlandia. En OA de rodilla y/o mano los principales trabajos fueron los de Reino Unido y el estudio GARP (Holanda). De esta forma se identificaron diversas regiones de ligamiento en las que se seleccionaron a posteriori los genes candidatos más probables para realizar estudios de asociación caso-control e identificar los polimorfismos asociados. De esta forma se identificaron como genes posiblemente implicados en OA MATN3, IL4-R y el cluster de IL-12-4. Sin embargo, la mayor parte de estos loci no han sido confirmados posteriormente. La dificultad en progresar desde ligamiento a identificación de genes de susceptibilidad se debe a que las regiones de ligamiento son normalmente muy extensas e identificar la variante de susceptibilidad es complicado. Además, los loci detectados tienen un efecto débil y el tamaño de las colecciones de muestras utilizadas fue pequeño, por lo que es posible que muchos hallazgos fuesen falsos positivos.
Estudios de genes candidatosLos estudios de genes candidatos toman como base de partida los conocimientos sobre la patogenia de la OA y seleccionan genes que por su papel funcional podrían ser relevantes en la susceptibilidad a la enfermedad. De esta forma, los genes candidatos estudiados en OA son genes que codifican para componentes de la matriz extracelular, para proteínas reguladoras del anabolismo o catabolismo de la matriz extracelular y para mediadores inflamatorios. Una vez seleccionados los genes, se realizan estudios de asociación caso-control en los que se comparan las frecuencias alélicas de los polimorfismos de interés, en individuos con OA no emparentados y controles libres de la enfermedad. Una de las características de estos estudios es que tienen la capacidad de detectar efectos pequeños con facilidad. Además, es más fácil obtener colecciones de muestras de un tamaño relativamente grande. Los estudios de genes candidatos detectaron mayoritariamente asociaciones que no se confirmaron posteriormente en los GWAS, como: PTGS-2, DIO-2 o FRZB5-7. Solo uno de los hallazgos de estos estudios, GDF-5, ha sido replicado posteriormente alcanzando el umbral de significación requerido en los GWAS8,9.
Estudios de asociación de genoma completoLos GWAS permiten estudiar toda la variabilidad genética del genoma sin hipótesis previa gracias a la existencia de desequilibrio de ligamiento (LD). La organización del genoma en bloques de LD permite obtener una representación de una región genómica seleccionando algunos SNPs informativos (tagSNPs). Los tagSNPs característicos de una región se conocen gracias al proyecto HapMap que estudió gran número de SNPs en diferentes poblaciones. Basándose en esta información, los arrays de genotipado de alta densidad proporcionan una cobertura casi completa de la variabilidad del genoma. Los arrays utilizados en la mayoría de los GWAS de OA contienen 500.000 SNPs y en el caso del estudio arcOGEN, el más potente realizado en OA, se utilizó un array que cubre 610.000 SNPs10,11. Una de las características principales de los GWAS es el umbral de significación considerado tras corregir por test múltiples (p<5×10−8). Este umbral viene dado por la estimación de que existen aproximadamente 106 SNPs independientes en el genoma humano (0,05/106=5×10−8) y tiene la ventaja de reducir la tasa de falsos positivos. Esta exigencia en el umbral de significación permite obtener resultados sólidos y, por tanto, replicables al precio de no descubrir otros factores genéticos con efecto más modesto. Este nivel de exigencia ha sido un gran estímulo para el desarrollo de estudios colaborativos y de metaanálisis para aumentar la potencia estadística a través del aumento del tamaño de las colecciones de muestras. La potencia de un GWAS depende además de la magnitud del efecto del SNPs causal y de la frecuencia de sus alelos. Gracias a los GWAS y a los metaanálisis de GWAS se han ido descubriendo genes de susceptibilidad a OA, aunque el número de loci es relativamente bajo en comparación con otras enfermedades complejas. Los primeros hallazgos sólidos fueron el gen GDF-5, el locus 7q22 y MCF2L9,12,13. Más recientemente se han incorporado los loci descubiertos en el estudio arcOGEN (GNL3, GLT8D1, ASTN2, FILIP1-SENP6, KLHDC-5-PTHLH, CHST11, TP63 y SUPT3H-CDC5L) y otros loci como DOT1L, NCOA3 y ALDH1A211,14-16. Se sospecha que la heterogeneidad de fenotipos de OA, el insuficiente tamaño de las colecciones de muestras disponibles y el mismo diseño de los GWAS pueden haber contribuido a estos resultados insuficientes. Todos estos factores se tratan en detalle a continuación.
Problemas específicos del estudio de la genética de la artrosisUno de los problemas que se plantea en el estudio genético de la OA es la heterogeneidad de fenotipos de la OA y cómo han sido considerados en los diferentes estudios que después se combinan en metaanálisis u otros estudios colaborativos. Esta variabilidad reduce el poder estadístico de los estudios de asociación. Por ello, recientemente se ha recomendado una estandarización de los fenotipos de los pacientes para futuros GWAS. Se recomienda estratificar los análisis por edad, sexo, BMI y presencia de OA radiográfica o sintomática. Dentro de la OA radiográfica (ROA) se deben definir los fenotipos en función de otra serie de características como el número de osteófitos (ROA de rodilla y mano) y la presencia de estrechamiento articular (ROA de cadera)17. Otra forma de aumentar el poder de detección de nuevas variantes es centrar los GWAS en el estudio de endofenotipos, que son fenotipos más próximos a la etiología biológica de la enfermedad que la enfermedad con su espectro de signos y síntomas. Se considera que los endofenotipos permiten obtener grupos de pacientes más homogéneos y tienen una relación más directa con los factores genéticos. De esta forma la potencia de los estudios puede aumentarse. El problema es la necesidad de obtener colecciones de muestras donde se hayan estudiado. En OA, los endofenotipos son características del cartílago o la forma de la articulación. Un ejemplo de endofenotipo es la anchura del espacio articular (JSW). Su utilización en un GWAS llevó al descubrimiento de un gen de susceptibilidad a OA de cadera, DOT1L14. Otro endofenotipo, el dolor en la articulación, ha permitido descubrir PACE-4 y TRPV1 como posibles loci asociados, aunque todavía no se ha llegado para ellos a una evidencia significativa a nivel de GWAS18,19.
Pero no solo la selección de los pacientes es importante, también la definición de los controles. La OA es una enfermedad de inicio tardío y de gran prevalencia en la población, por lo que el uso de controles poblacionales sin seleccionar es muy discutido. Si los casos que se incluyen en los estudios genéticos están seleccionados por criterio radiográfico sería ideal que también lo estuvieran los controles. Sin embargo, el diseño de los estudios no siempre lo permite debido a la dificultad que representa en muchas ocasiones justificar una exploración radiográfica en individuos sanos. En la primera fase del estudio arcOGEN se evaluó cómo influye utilizar controles poblacionales o controles libres de OA en los resultados de los GWAS. Los controles libres de OA de este estudio, también denominados «supercontroles», son controles de la colección de gemelos de Twins UK, libres de OA y seleccionados radiográficamente según una escala de Kellgren-Lawrence<2. De todos los loci de susceptibilidad para OA analizados en el estudio de arcOGEN, solo GDF-5 mostró un efecto mayor y un valor de p más significativo al utilizar controles libres de OA. En el resto de los SNPs analizados se obtuvieron resultados idénticos o en algunos casos mejores, al utilizar los controles poblacionales. Debido a ello, se concluyó que utilizar controles libres de OA no produce una mejoría detectable en los resultados de asociación. Sin embargo, este resultado no puede considerarse definitivo, ya que muchas de estas asociaciones se habían descubierto utilizando controles poblacionales20.
Las poblaciones incluidas en el estudio son otra fuente de heterogeneidad. Se ha comprobado que un mismo factor genético no tiene igual efecto en la susceptibilidad a la enfermedad en distintas poblaciones. Esto se refleja claramente en los estudios genéticos realizados en poblaciones europea y asiática. Así, las asociaciones a nivel de GWAS de HLA de clase ii/iii y DVWA son específicas de población japonesa y no se replicaron en diferentes estudios realizados en población europea. También hay que tener en cuenta que los factores genéticos de susceptibilidad son específicos de articulación (OA de rodilla, cadera y mano) y de sexo. Esta compartimentalización dificulta reunir colecciones de muestras de gran tamaño. Precisamente, el tamaño de las colecciones disponibles en OA es también un factor limitante. Por ejemplo, el metaanálisis que identificó la asociación del locus 7q22 con OA de rodilla incluía un total de 6.709 casos y 35.909 controles. Sin embargo, el poder estadístico de este estudio para descubrir variantes de efectos pequeños (OR=1,10-1,15) no es suficiente. Se necesitarían 7.000 muestras más de OA de rodilla para detectar el efecto de SNPs con una frecuencia del alelo menor (MAF)=20% y una OR de 1,15. Luego, las colecciones de muestras disponibles no son suficientes para identificar efectos modestos.
La mayor parte de los estudios de asociación en artrosis son estudios de asociación caso-control, aunque también se han realizado estudios de cohortes. En ellos se hace un seguimiento de un grupo representativo de la población a lo largo del tiempo evaluándose la aparición de nuevos casos de OA. Las características positivas de estos estudios son su representatividad, el control de la recogida de la información según protocolos establecidos y la ausencia de sesgos, ya que los parámetros demográficos y ambientales están claramente definidos y son comunes a los casos y los controles. Sin embargo, su principal limitación es la dificultad de iniciar el estudio con un gran número de individuos a los que hay que realizar un seguimiento a lo largo de muchos años para conseguir un número suficiente de casos.
Por otra parte, los GWAS tienen otras limitaciones que vienen dadas por la cobertura incompleta proporcionada por los arrays de genotipado. Los SNPs con frecuencia <5%, que presentan un LD muy débil con otros SNPs, no se estudian de manera fiable. Esta falta de cobertura es más marcada para las variantes genéticas raras (con MAF<0,5%), algunas variantes de número de copias (VNTR) complejas, y los microsatélites que tienen múltiples alelos y un alto nivel de mutación en la población21. Otra de las limitaciones de los GWAS, esta vez en la interpretación de los resultados, es que los SNPs asociados no tienen por qué ser las variantes causales de la enfermedad. En los últimos años se ha desarrollado la tecnología que permite resecuenciación del genoma, o de la parte del genoma que se expresa como proteínas (el exoma), y es posible que de esta forma se pueda recuperar toda la información perdida en los GWAS con arrays de SNPs y que sea más fácil identificar los polimorfismos causales del aumento de susceptibilidad. Este último objetivo también se ha facilitado con el proyecto ENCODE (ENCyclopedia Of DNA elements), que proporciona información sobre la funcionalidad de todos los segmentos del genoma humano22.
En resumen, se pueden considerar 2 grandes grupos de loci asociados con OA:
- 1
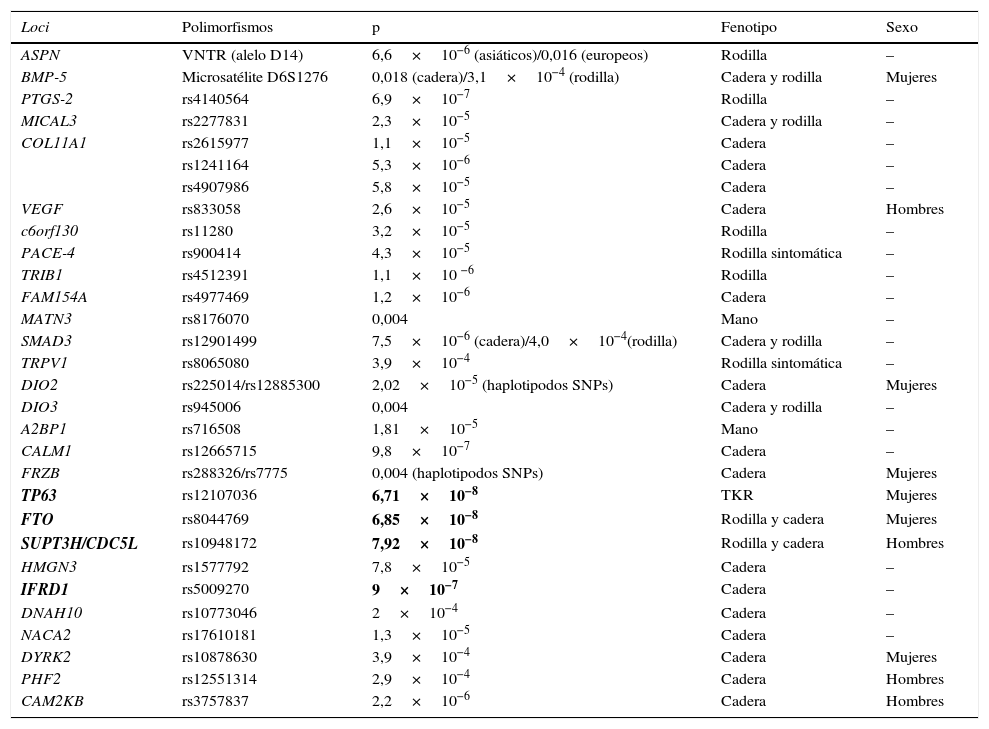
Loci posiblemente asociados con OA pero que no han alcanzado el nivel de asociación requerido en los GWAS (tabla 1).
Tabla 1.Loci asociados con artrosis que no alcanzan el nivel de asociación requerido en los GWAS
Loci Polimorfismos p Fenotipo Sexo ASPN VNTR (alelo D14) 6,6×10−6 (asiáticos)/0,016 (europeos) Rodilla – BMP-5 Microsatélite D6S1276 0,018 (cadera)/3,1×10−4 (rodilla) Cadera y rodilla Mujeres PTGS-2 rs4140564 6,9×10−7 Rodilla – MICAL3 rs2277831 2,3×10−5 Cadera y rodilla – COL11A1 rs2615977 1,1×10−5 Cadera – rs1241164 5,3×10−6 Cadera – rs4907986 5,8×10−5 Cadera – VEGF rs833058 2,6×10−5 Cadera Hombres c6orf130 rs11280 3,2×10−5 Rodilla – PACE-4 rs900414 4,3×10−5 Rodilla sintomática – TRIB1 rs4512391 1,1×10 −6 Rodilla – FAM154A rs4977469 1,2×10−6 Cadera – MATN3 rs8176070 0,004 Mano – SMAD3 rs12901499 7,5×10−6 (cadera)/4,0×10−4(rodilla) Cadera y rodilla – TRPV1 rs8065080 3,9×10−4 Rodilla sintomática – DIO2 rs225014/rs12885300 2,02×10−5 (haplotipodos SNPs) Cadera Mujeres DIO3 rs945006 0,004 Cadera y rodilla – A2BP1 rs716508 1,81×10−5 Mano – CALM1 rs12665715 9,8×10−7 Cadera – FRZB rs288326/rs7775 0,004 (haplotipodos SNPs) Cadera Mujeres TP63 rs12107036 6,71×10−8 TKR Mujeres FTO rs8044769 6,85×10−8 Rodilla y cadera Mujeres SUPT3H/CDC5L rs10948172 7,92×10−8 Rodilla y cadera Hombres HMGN3 rs1577792 7,8×10−5 Cadera – IFRD1 rs5009270 9×10−7 Cadera – DNAH10 rs10773046 2×10−4 Cadera – NACA2 rs17610181 1,3×10−5 Cadera – DYRK2 rs10878630 3,9×10−4 Cadera Mujeres PHF2 rs12551314 2,9×10−4 Cadera Hombres CAM2KB rs3757837 2,2×10−6 Cadera Hombres TKR: reemplazamiento articular total de rodilla.
Se resaltan en negrita los loci que se encuentran cerca del umbral de asociación de los GWAS (p<5×10−8) en europeos. Estos loci proceden del estudio arcOGEN y del reciente metaanálisis de OA de cadera. Evangelou et al15 hace referencia al metaanálisis de OA de cadera no a la tabla
- 2
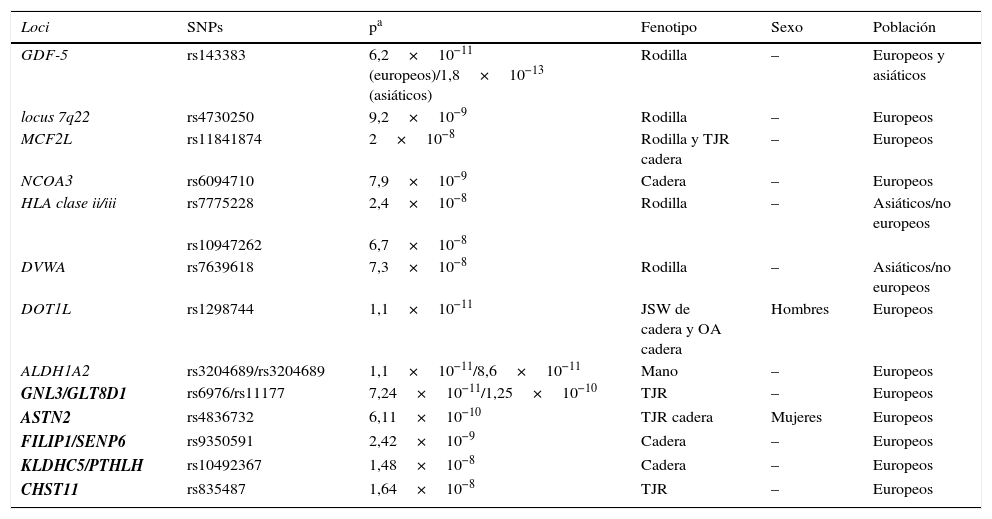
Loci que han alcanzado el nivel de asociación de los GWAS (p<5×10−8) y que, por lo tanto, son los más consistentemente asociados con OA (tabla 2).
Tabla 2.Loci asociados en artrosis que alcanzan el nivel de asociación requerido en los GWAS
Loci SNPs pa Fenotipo Sexo Población GDF-5 rs143383 6,2×10−11 (europeos)/1,8×10−13 (asiáticos) Rodilla – Europeos y asiáticos locus 7q22 rs4730250 9,2×10−9 Rodilla – Europeos MCF2L rs11841874 2×10−8 Rodilla y TJR cadera – Europeos NCOA3 rs6094710 7,9×10−9 Cadera – Europeos HLA clase ii/iii rs7775228 2,4×10−8 Rodilla – Asiáticos/no europeos rs10947262 6,7×10−8 DVWA rs7639618 7,3×10−8 Rodilla – Asiáticos/no europeos DOT1L rs1298744 1,1×10−11 JSW de cadera y OA cadera Hombres Europeos ALDH1A2 rs3204689/rs3204689 1,1×10−11/8,6×10−11 Mano – Europeos GNL3/GLT8D1 rs6976/rs11177 7,24×10−11/1,25×10−10 TJR – Europeos ASTN2 rs4836732 6,11×10−10 TJR cadera Mujeres Europeos FILIP1/SENP6 rs9350591 2,42×10−9 Cadera – Europeos KLDHC5/PTHLH rs10492367 1,48×10−8 Cadera – Europeos CHST11 rs835487 1,64×10−8 TJR – Europeos JSW: anchura del espacio articular; TJR: reemplazamiento articular total.
En negrita se marcan los loci del estudio arcOGEN.
En este apartado se tratan los loci asociados con OA más relevantes.
GDF-5, el único gen candidato que se ha confirmadoGDF-5 fue el primer locus asociado a nivel de GWAS en europeos y asiáticos. Un estudio en población japonesa descubrió la asociación de un SNPs (rs143383, C/T) en la región 5′UTR del gen. Esta asociación se observó con OA de cadera (p=1,8×10−13) y con menor magnitud con OA de rodilla8. Estudios posteriores confirmaron la asociación de rs143383 en asiáticos y en europeos. Además, en un metaanálisis de cohortes europeas y asiáticas se encontró asociación de rs143383 a nivel de GWAS con OA de rodilla (p=6.2×10−11)9,23. Se debe destacar que este mismo SNPs se encontró asociado con diversos fenotipos más o menos relacionados con OA como displasia de cadera, tamaño óseo, riesgo de fractura vertebral en mujeres y con la altura24,25. En relación con el papel funcional de este gen, ya era bien conocido que GDF-5 es un miembro de la familia de las proteínas morfogenéticas del hueso (BMP) que tiene una participación clave en el desarrollo del esqueleto, la condrogénesis, la formación de las articulaciones diartroideas, y también en los procesos de reparación del hueso y del cartílago26,27. El SNPs rs143383 influye en la actividad transcripcional de GDF-5 con uno de sus alelos, el alelo T asociado con OA, mostrando una expresión disminuida en el cartílago. Con posterioridad se han identificado otros 2 polimorfismos en GDF-5 que modifican su expresión y que contribuyen a su asociación con OA, ya que están en LD con rs14338323. La expresión de GDF-5 también está modificada por una serie de reguladores en trans que interactúan con el rs143383 en la región 5′UTR y además es un locus sujeto a regulación epigenética28,29. Sin duda, GDF-5 es el factor genético más consistentemente demostrado en los estudios genéticos de OA y con un papel funcional más claramente establecido en la biología del cartílago articular.
El locus del cromosoma 7q22 con sus 6 genesEl segundo locus que se encontró asociado a nivel de GWAS en europeos es el locus del cromosoma 7q22. Es un locus muy extenso que ocupa unas 500kb, y contiene 400 SNPs y 6 genes diferentes: PRKAR2B, HBP1, COG5, GPR22, DUS4L y BCAP29. La asociación de este locus con OA de rodilla se descubrió en un GWAS que después de diferentes fases de estudio de 500.000 SNPs llevó a solo un SNPs, rs3815148, con asociación a nivel de GWAS (OR=1,14, p=8×10−8)10. Posteriormente se realizó un metaanálisis con los datos de 4 GWAS de población europea seguido de una replicación en 9 colecciones europeas adicionales. En este estudio, rs4730250 (DUS4L) fue el SNPs más asociado tanto en la fase inicial como en el análisis global de todas las colecciones (OR=1,17, p=9,2×10−9)30. Los estudios funcionales del locus se centraron en descubrir cuál de los 6 genes podría ser el mejor candidato funcional. En principio todo parecía indicar que era GPR22, debido a que un SNPs localizado antes del gen y en elevado ligamiento con rs3815148 se asoció con cambios en la expresión del gen en linfoblastos. Sin embargo, su papel ha sido cuestionado con posterioridad porque parece que no se expresa casi en condrocitos humanos en cultivo y tampoco en muestras de cartílago. En contraste con estos datos, la inmunohistoquímica muestra expresión de GPR22 en los condrocitos de la capa superficial de ratones con OA, y la sobreexpresión de GPR22 in vitro estimula el proceso hipertrófico de los condrocitos y acelera la calcificación31. Experimentos más recientes han priorizado HBP1 como el mejor gen para explicar la asociación. Se llegó a esta conclusión analizando la expresión alélica diferencial de los 6 genes en cartílago de pacientes con OA. Este gen es importante en la ruta de señalización Wnt32. Sin embargo, esta cuestión no puede considerarse resuelta y nadie ha propuesto todavía un mecanismo detallado como el descrito para GDF-5.
El estudio arcOGEN (fases 1 y 2) y sus 5 lociEl proyecto arcOGEN es el más ambicioso realizado hasta el momento en el campo y constituye un gran esfuerzo para el esclarecimiento de las bases genéticas de susceptibilidad a la OA. En la primera fase se estudiaron 3.177 casos (OA de rodilla y cadera) y 4.894 controles del Reino Unido. Esta fase llevó a la identificación de algunos loci asociados, ninguno de ellos a nivel de GWAS y ninguno de ellos confirmado con posterioridad. Sin embargo, esta fase de arcOGEN fue utilizada para analizar la estructura genética de la OA, y este resultado sí que es interesante. Esta estructura es la de una enfermedad poligénica, como la de la mayoría de las enfermedades complejas, y no oligogénica, como se había postulado antes del inicio de los GWAS. Esto significa que muy probablemente hay muchos polimorfismos asociados, cada uno de ellos con un efecto pequeño en la predisposición a la enfermedad y que además están repartidos de una forma homogénea por todo el genoma. En contraste con otras enfermedades complejas en las que muchos polimorfismos con pequeño efecto se encuentran junto a unos pocos de efecto marcado, en la OA no se ha encontrado ninguna evidencia de polimorfismos de gran efecto.
En la segunda fase de arcOGEN (con un total de 7.410 casos con OA de rodilla y de cadera, y 11.009 controles) se identificaron 5 loci con asociación a nivel de significación de GWAS y otros 3 alcanzando un nivel muy cercano. Constituye, por tanto, el estudio con el que más se ha avanzado en la identificación de factores genéticos de susceptibilidad para la OA. Estos resultados se obtuvieron combinando el GWAS de las muestras de arcOGEN con la replicación in silico en un número similar de casos y controles de 6 colecciones europeas y la replicación de novo en otra cohorte del Reino Unido. Los 2 SNPs más asociados, rs6976 y rs11177, representan una única señal que está asociada con OA y reemplazamiento articular tanto de la rodilla como de la cadera. Uno de estos SNPs, rs6976, está situado en la región 3′UTR de GLT8D1, y el otro, rs11177, es un SNPs no sinónimo localizado en el exón 3 de GNL3, por lo que cualquiera de estos genes podría ser el responsable de la asociación. Los otros 4 loci que alcanzaron nivel de GWAS estaban asociados con OA de cadera y se han descrito como: ASTN2, FILIP1-SENP6, KLHDC5-PTHLH y CHST11, aunque todavía no se han realizado estudios que permitan atribuir causalidad a ninguno de estos genes. Los 3 loci que mostraron asociación cerca del umbral de p<5×10−8 se han descrito como: TP63, FTO y SUPT3H-CDC5L11(tabla 2). Se debe señalar que, al igual que en la primera fase de arcOGEN, no se replica la asociación de GDF-5 ni del locus 7q22 a nivel de GWAS, a pesar de que, como se ha señalado, se trata de asociaciones sólidas. Esta falta de replicación se ha atribuido a una combinación de diferentes factores: insuficiente poder estadístico dado el tamaño muestral en la fase de descubrimiento y las frecuencias alélicas de los SNPs y, en GDF-5, la ausencia de genotipado de rs143383. Sin embargo, un tagSNPs de rs143383, el rs4911494 (r2=0,94), tampoco mostró asociación. De algunos de los loci asociados en la segunda fase de arcOGEN, como ASTN2, se desconoce cuál puede ser su función específica en el cartílago articular, si es que la tienen. De otros se sabe algo más, como de GNL3, que mostró expresión en las células mesenquimales, precursoras de los condrocitos, y en condrocitos artrósicos. Cerca de los genes FILIP1-SENP6 se encuentra COL12A1, que codifica para una proteína de colágeno que forma parte del entramado de fibras de la matriz extracelular. En relación con otro locus, el gen PTHLH codifica la proteína relacionada con la hormona paratiroidea (PTHrP), que muestra una expresión elevada en el cartílago de pacientes con OA y participa en el desarrollo del hueso subcondral. CHST11, por su parte, codifica una proteína implicada en la síntesis de proteoglicanos, concretamente del sulfato de condroitina. El sulfato de condroitina no solo es un componente del cartílago sino que se utiliza como tratamiento de la OA, aunque su efectividad es discutida33. Otro de los posibles genes, TP63, está relacionado con el desarrollo de la articulación y se ha descrito como uno de los loci que determinan la morfología facial en humanos34. Por su parte, cerca del locus CDC5L-SUPT3H se encuentra RUNX-2, que es esencial tanto en la diferenciación de los osteoblastos como en la morfogénesis del esqueleto. Ninguno de los loci anteriores ha sido todavía estudiado más allá de su descripción como asociado. El único que ha sido objeto de un estudio posterior es el locus caracterizado por contener al gen FTO. Este gen se había asociado con anterioridad con obesidad y se había demostrado que predisponía al aumento de peso por mecanismos no totalmente aclarados pero que incluirían regulación de la ingesta o de la producción de energía. Esto hizo sospechar que la asociación con la OA fuese secundaria a la asociación con obesidad, ya que la obesidad es un factor de riesgo importante para la OA, especialmente para la OA de rodilla. Al analizar esta posibilidad se observó que la asociación de FTO con OA desaparecía tras ajustar por el BMI, lo que vuelve a sugerir una etiología genética común y resalta la importancia de la estratificación por BMI en los estudios genéticos de OA35.
COL11A1De entre las asociaciones propuestas en la primera fase de arcOGEN y no confirmadas con posterioridad, el gen COL11A1 es el más prometedor. Esto se debe al papel funcional de este gen que codifica para una de las 3 fibrillas que forman la molécula de colágeno de tipoxi. Este colágeno se une a los agregados de proteoglicanos y los ancla a la red de colágeno de la matriz extra celular36. Pero también resalta porque algunas mutaciones en este gen dan lugar a síndromes hereditarios que incluyen en su espectro de manifestaciones OA de inicio temprano37. Una evidencia más indirecta es su asociación con hernia de disco lumbar (LDH) en japoneses, ya que la biología del disco intervertebral y del cartílago articular están relacionadas. La expresión de COL11A1 disminuye en el disco intervertebral de pacientes con LDH y con el alelo de susceptibilidad a LDH (aleloC) del SNPs asociado rs167648638. Sin embargo, rs1676486 no está asociado con OA, aunque sí lo está con la expresión de COL11A1 en cartílago artrósico. Por si esto no fuera suficientemente paradójico, el SNPs de COL11A1 más asociado con OA en el estudio arcOGEN fase 1, rs2615977, no muestra ninguna asociación con la expresión del gen en el cartílago39.Es posible que un reciente metaanálisis de 9 GWAS de artrosis haya proporcionado pistas para poder explicar estos resultados, ya que mostró asociación independiente de 2 SNPs de COL11A1 con OA de cadera localizados en los extremos del gen40. Por lo tanto, es posible que las paradojas de los estudios previos se resuelvan al considerar la posibilidad de 2 asociaciones independientes, ninguna de ellas completamente coincidente con las estudiadas funcionalmente hasta ahora.
DOT1L y artrosis de caderaComo ya se ha comentado, la asociación de DOT1L se descubrió al estudiar un endofenotipo de OA de cadera, la anchura del espacio articular (JSW). Aunque se ha utilizado como un ejemplo de la eficacia del uso de endofenotipos, hay que hacer notar que es el único locus descubierto en el GWAS que lo usó, lo que no deja de ser un rendimiento pobre para un estudio como el de la cohorte de Rotterdam (6.523 sujetos) analizando una variable cuantitativa (que normalmente proporciona más potencia que una variable dicotómica). Con posterioridad se ha demostrado que el mismo SNPs de DOT1L está asociado con OA de cadera en hombres (p=7,8×10−9) en un estudio con elevado tamaño muestral. DOT1L es una metiltransferasa de histonas que interactúa con TCF-4 regulando la transcripción de los genes de la ruta Wnt e influenciando la diferenciación condrogénica, se expresa en el cartílago de los pacientes con OA y su supresión inhibe la acumulación de proteoglicanos y de cartílago durante la condrogénesis14.
MCF2L ayudado por los estudios de resecuenciación del genomaOtro análisis reciente descubrió la asociación de MCF2L con OA de cadera. Este estudio utilizó la información del Proyecto 1000 Genomas para obtener la suficiente confianza para afirmar esta asociación. El Proyecto 1000 Genomas pretende proporcionar un catálogo completo de toda la variación genética que alberga el genoma humano. Mediante secuenciación de última generación seguida de distintas etapas de genotipado y validación, se catalogan variantes comunes, variantes de baja frecuencia (0,5-5%) y variantes raras (<0,5%). Esta información se utilizó para imputar SNPs y variantes raras que no se habían genotipado en el GWAS de la fase1 de arcOGEN. La comparación entre casos y controles de más de 7 millones de variantes con frecuencia >1% llevó a seleccionar 8 SNPs en 6 loci para genotipar directamente en las muestras del GWAS original. De esta forma se encontraron nuevos SNPs asociados en MCF2L, donde solo se había encontrado rs11841874, por lo que se había considerado un resultado dudoso y no se había proseguido su investigación. Una vez que se aumentó la confianza en este resultado se analizó en otras cohortes, llevando a una asociación significativa a nivel de GWAS (p metaanálisis=2×10−8). Poco se sabe sobre el papel funcional de MCF2L más allá de que participa en la regulación de la neurotrofina-3 que pertenece a la familia NGF, un factor pro-angiogénico cuya expresión se encuentra aumentada en condrocitos artrósicos13.
NCOA3 y artrosis de caderaEn un metaanálisis de GWAS de OA de cadera se descubrió la asociación del rs6094710 (p=7,9×10−9). Este estudio reunió un total de 4.349 casos de OA de cadera y 46.903 controles en la fase de descubrimiento y 11.277 casos y 67.473 controles en la replicación, por lo que es el mayor realizado hasta ahora. El SNPs rs6094710 se encuentra cerca de NCOA3, que es un gen con expresión disminuida en cartílago artrósico. Además, rs6094710 está en LD con un SNPs no sinónimo (Arg>Cys) que es probablemente dañino para una variante de la proteína, lo que apunta a un posible mecanismo molecular. NCOA3 podría tener un papel en el metabolismo del hueso, ya que es un coactivador de varios receptores nucleares como los de retinoides, de la vitaminaD o de la hormona tiroidea (T3). Aunque también es posible que NCOA3 participe en el proceso de mecanotransducción de los condrocitos15.
DVWA y HLA de clase ii/iii y artrosis en asiáticosLos loci DVWA y HLA de clase ii/iii muestran asociación a nivel de GWAS pero restringida a población asiática. Son 2 ejemplos de la heterogeneidad genética existente entre europeos y asiáticos. La asociación de DVWA con OA de rodilla se identificó en un GWAS en población japonesa41. Con posterioridad se han realizado 2 estudios específicos de los mismos polimorfismos en población europea, pero ninguno de ellos detectó asociación42,43. La función de este gen ha sido muy poco estudiada. En el trabajo que descubrió su asociación se comprobó que DVWA interactúa con la β-tubulina de manera diferencial dependiendo de los alelos de los 2 SNPs no sinónimos que habían mostrado asociación. Posteriormente ha habido controversia sobre la naturaleza de este gen. Así, se encontraron evidencias de que DVWA era en realidad la porción 5′ de otro gen, COL6A444. Sin embargo, la reciente clonación del gen parece haber encontrado una explicación, ya que reveló 2 isoformas de la proteína, una larga y otra corta. La corta no tiene homología con COL6A4 y tiene una expresión específica en cartílago. Se ha hipotetizado el posible papel de DVWA en la diferenciación de los condrocitos y el tráfico intracelular45.
La asociación de HLA se descubrió en un GWAS realizado en población japonesa con OA de rodilla. Había 2 SNPs asociados: rs7775228 en HLA-DQ1B y rs10947262 en BTNL-2 (p=2,4×10−8 y p=6,7×10−8, respectivamente) que están localizados en la región del HLA de clase ii/iii y que no son independientes, representando, por tanto, una única asociación46. Estos SNPs no han estado asociados con OA de rodilla en muestras europeas en el mismo estudio o en estudios posteriores47,48. La importancia de esta asociación reside en que podría explicar parte del componente inflamatorio de la OA.
ALDH1A2 y artrosis de manoHasta la fecha, A2BP1 se consideraba el locus más relevante en OA de mano y el rs716508 el SNPs más fuertemente asociado con este fenotipo (p=1,81×10−5)49. Sin embargo, recientemente se ha descubierto una nueva asociación a nivel de GWAS con OA de mano16. En la fase de descubrimiento del estudio, realizada en población islandesa, se encontró un grupo de 55 variantes asociadas (p<5×10−8). Todas ellas se encuentran en un bloque de LD en el cromosoma 15q22 y se clasificaron en 2 grupos considerando la frecuencia del alelo de riesgo (41 y 52%) con 2 SNPs representativos para cada grupo, el rs4238326 y el rs3204689. Las variantes de ambos grupos se testaron en 5 colecciones muestrales europeas adicionales, y el análisis conjunto de ambas fases mostró asociación a nivel de GWAS para el rs4238326 (OR=1,44; p=8,6×10−11) y el rs3204689 (OR=1,46; p=1,1×10−11). En relación con el papel funcional del locus, ALDH1A2 codifica para la retinaldehído deshidrogenasa 2, que cataliza la síntesis de ácido retinoico, molécula con papel relevante en el desarrollo del cartílago y el hueso.
El papel del ADN mitocondrial en la artrosisAdemás del genoma nuclear también se ha estudiado el posible papel de los haplogrupos del ADNmt en OA. Cada haplogrupo mitocondrial está definido por una combinación particular de variantes genéticas. En concreto, los haplogrupos J (m.4216T>C,m.10398A>G) y JT (m.4216T>C) se encontraron asociados con un menor riesgo de padecer OA de rodilla y los haplogrupos relacionados J y J1c (m.14798T>C) con un menor riesgo de OA de cadera en españoles50,51. En población asiática también se encontró asociación de otros 2 haplogrupos mitocondriales, G y B/B4, que mostraron un efecto de susceptibilidad y protección, respectivamente52. Sin embargo, en el estudio arcOGEN de un tamaño muestral mayor (7.393 casos y 5.122 controles del Reino Unido) no se encontró asociación con ningún haplogrupo de ADNmt53. Se ha sugerido que diferencias en la frecuencia de los haplogrupos en la población de los diferentes estudios (España y Reino Unido) podrían justificar las discrepancias, además de que la falta de replicación en este estudio no constituye una evidencia suficiente, como ya se ha mencionado en relación con los loci GDF-5 y 7q2254. Se han encontrado otras asociaciones que podrían reforzar la implicación de los haplogrupos mitocondriales en la patogénesis de la OA, como la asociación con los niveles en suero de colágeno de tipoii y metaloproteasas55.
Futuro de los estudios genéticos de artrosisEl estudio de los factores genéticos implicados en la susceptibilidad a la OA es un campo que ha progresado notablemente en los últimos años. En la actualidad se puede hablar de 11 loci asociados con OA en población europea al nivel requerido en los GWAS y 4 cerca de este nivel. Aun así, el número de genes de susceptibilidad encontrados es relativamente bajo si lo comparamos con otras enfermedades complejas. Los investigadores del campo proponen una serie de estrategias adicionales para aplicar en estudios futuros que ayudarían a completar nuestro conocimiento sobre el componente genético de la OA. Entre ellas se incluyen aumentar el tamaño de los estudios, estandarizar los fenotipos de los pacientes y utilizar la información del Proyecto 1000 Genomas para completar la cobertura de la variación genética. También puede ser útil realizar otros estudios, como los de resecuenciación del genoma, o del exoma, en búsqueda de variantes raras con elevada penetrancia y el estudio de microsatélites y VNTR, que son variantes no estudiadas en los GWAS. Además, también se podría extraer más información de los GWAS ya realizados con nuevas estrategias de estudio, como han sido los metaanálisis de genes candidatos y pueden ser los análisis basados en genes.
En relación con la búsqueda de variantes raras, podría ser de gran interés secuenciar genes ya asociados, pues estos genes pueden portar variantes raras asociadas, como se ha observado en otras enfermedades. Sin embargo, un estudio de este tipo del gen GDF-5 no encontró ninguna variante rara que contribuya a la susceptibilidad a OA56.
En relación con los microsatélites y VNTR, hay 2 que han mostrado asociación con OA en algunos estudios: un VNTR en ASPN (asporina) y un microsatélite en el intrón1 de BMP-5. La asociación del VNTR de ASPN como un factor genético de susceptibilidad a la OA se detectó en población japonesa. La frecuencia del alelo de 14 repeticiones del VNTR (D14) se encontró elevada en los pacientes con OA de rodilla respecto a los controles (p=0,000066). La repetición D13 mostró un efecto menor y de sentido contrario o protector, esto es, que su frecuencia estuvo elevada en controles57. Esta asociación no se encontró en población europea, lo que vuelve a demostrar la existencia de un componente étnico diferencial entre ambas poblaciones. Por su parte, el microsatélite de BMP-5, D6S1276, mostró asociación (p=0,018) con OA de cadera en mujeres del Reino Unido. Además, se mostró que afectaba la actividad transcripcional del gen in vitro. Este hallazgo motivó estudios posteriores de BMP-5, demostrándose su asociación en otro fenotipo, OA de rodilla en pacientes con artrosis de 3 países europeos58,59. Dado que ni la asociación de ASPN ni la de BMP-5 han alcanzado el nivel de GWAS, no se les ha prestado demasiada atención, pero sugieren que las variantes en el número de copias pueden esconder factores genéticos de OA.
Otra de las aproximaciones mencionadas trata de extraer más información de los GWAS ya realizados. Ya hay gran experiencia demostrando el valor de los metaanálisis. Un complemento es el estudio centrado en conjuntos particulares de genes de los cuáles existe algún tipo de evidencia independiente de participación en la OA. Una aproximación de este tipo ya se ha realizado utilizando como fuente de la evidencia previa los estudios de genes candidatos. Este análisis resaltó la posible asociación con OA de cadera de COL11A1 y VEGF40. Otra estrategia que podría ser útil es la de obtener un estadístico combinando las asociaciones independientes de los SNPs de un gen. Esto es lo que se ha denominado Gene-Based Analysis (GBA). Se basa en la hipótesis de que múltiples variantes genéticas en genes importantes para la enfermedad podrían contribuir, aunque ninguno lo haga con un nivel suficiente para ser detectado a nivel individual. Se han realizado algunos estudios de este tipo en otras enfermedades, pero sin resultados muy convincentes. Es posible que para poder realizar este tipo de análisis se requieran nuevas herramientas.
Por lo tanto, aunque los últimos años han traído muchos éxitos en la identificación de loci asociados con enfermedades complejas, los investigadores de la OA no estamos satisfechos, pues somos conscientes de que quedan muchos más por identificar. Se ha planteado toda una serie de aproximaciones que pueden ayudarnos a avanzar más en este camino, aunque todavía no está claro cuál de ellas será más fructífera. Es posible, que cada una nos proporcione algunos avances y que solo poco a poco se vaya completando nuestro conocimiento del componente genético de la OA. Otro aspecto en el que hasta ahora solo se han dado los primeros pasos es la identificación de las variantes causales del aumento de susceptibilidad y de los mecanismos sobre los que tienen un efecto. Esta es un área que requiere un desarrollo urgente, pues es donde existen más posibilidades de contribuir al conocimiento y manejo de la enfermedad.
Responsabilidades éticasDerecho a la privacidad y consentimiento informadoLos autores declaran que en este artículo no aparecen datos de pacientes.
Confidencialidad de los datosLos autores declaran que en este artículo no aparecen datos de pacientes.
Protección de personas y animalesLos autores declaran que para esta investigación no se han realizado experimentos en seres humanos ni en animales.
Conflicto de interesesLos autores declaran no tener ningún conflicto de intereses.